Biomodelli computazionali

 GENOMICA COMPUTAZIONALE
GENOMICA COMPUTAZIONALE
POLIMI: sviluppato il GenoMetric Query Language (GMQL), un innovativo sistema per l’analisi di big data genomici che si colloca all’avanguardia mondiale della ricerca di settore
23.06.2018
Testo dell’articolo
GeCo ha un obiettivo rivoluzionario: sviluppare e consolidare un nuovo approccio alla medicina integrando l’analisi di big data derivanti dal sequenziamento del genoma per trovare una risposta più precisa ed efficace a tante domande della biologia e della medicina, incluse la modalità di sviluppo dei tumori e la loro dipendenza da cause ambientali. GeCo vuole rivisitare la genomica computazionale tramite l’uso estensivo di banche dati pubbliche, ideando nuovi modelli, linguaggi e strumenti per la loro analisi e gestione, solidi dal punto di vista dei concetti utilizzati e capaci di operare in modo super-efficiente su sistemi cloud.
L’equipe del progetto GeCo ha sviluppato il sistema GMQL per interrogare dati genomici scaricati da grandi banche dati prodotte da Consorzi internazionali, un risultato che si colloca all’avanguardia mondiale della ricerca di settore. GMQL è un sistema aperto, utilizzabile pubblicamente, ed il Broad Institute (USA) lo ha reso disponibile ai suoi ricercatori.
Il Broad Institute è un centro di ricerca di eccellenza per la Genomica Computazionale e lo studio delle malattie, con particolare enfasi sui tumori e sulle malattie cardio-vascolari, infettive e psichiatriche; coinvolge ricercatori provenienti da Harvard, MIT e da diversi ospedali legati ad Harvard, che operano in discipline diverse, tra cui la medicina, la biologia, la chimica, l’informatica, la matematica e l’ingegneria.
Il gruppo di Data Science del Broad Institute è responsabile di FireCloud, una piattaforma aperta per l’analisi dei dati genomici che garantisce sicurezza ed elevate prestazioni; FireCloud consente l’accesso protetto a dati genomici presenti in TCGA (The Cancer Genome Atlas) e l’uso di GATK, un software sviluppato al Broad Institute per l’individuazione di mutazioni. Al termine di un percorso di integrazione del software, anche GMQL è disponibile su FireCloud. Usando una workspace pubblica creata per GMQL, i ricercatori possono vedere GMQL al lavoro su tre casi di studio di progressiva complessità, e possono quindi utilizzare il codice sviluppato al Politecnico di Milano per integrare e completare le loro analisi dei dati. Il gruppo del Politecnico ha fatto da apripista nella sperimentazione dell’uso delle workspace pubbliche, uno strumento di FireCloud per facilitare l’integrazione con strumenti software sviluppati da gruppi di ricerca esterni al Broad Institute.
Nel corso del progetto, il sistema sarà arricchito di strumenti per l’analisi dei dati e verrà reso sempre più efficiente, utilizzando vari framework per la gestione di dati disponibili su server paralleli e in ambiente cloud. Tra gli obiettivi del progetto vi è anche la costruzione di un open source messo a disposizione dei ricercatori biologici e clinici, che potranno usare servizi offerti dal sistema oppure scaricarlo e installarlo presso i loro centri. Mentre i servizi realizzati dal Politecnico di Milano useranno esclusivamente dati pubblici, messi a disposizione per uso secondario, cioè per attività di ricerca, l’installazione protetta del sistema in un contesto clinico potrà essere utilizzata per la cosiddetta “medicina personalizzata”, cioè l’adattamento delle terapie ai dati genomici di specifici pazienti. L’obiettivo più ambizioso di GeCo è la realizzazione di un Internet per la genomica, cioè di un modo di raccogliere dati genomici pubblicati da consorzi internazionali e dai ricercatori, e di un Google per la genomica, cioè un sistema di indicizzazione e ricerca su grandi raccolte di dati genomici pubblici. Questi strumenti potranno essere usati per facilitare in futuro lo studio approfondito di gravi malattie.
Cos’è la Genomica Computazionale
É la scienza che, partendo dal sequenziamento del genoma e grazie all’uso di analisi statistiche e computazionali, decifra la funzione delle regioni del genoma e costituisce pertanto il presupposto per le future scoperte nel campo della biologia e della medicina. Le tecniche di sequenziamento del genoma di nuova generazione NGS (Next Generation Sequencing) consentono oggi la produzione dell’intera sequenza del genoma umano a costi molto bassi. Parallelamente sono stati sviluppati algoritmi specializzati per estrarre le caratteristiche salienti del genoma che si vuole studiare, per evidenziare ad esempio le mutazioni o l’espressione dei geni, cioè la loro attività di trascrizione. La grande lacuna da colmare rimane però l’ideazione di un sistema capace di integrare i dati genomici estratti da tali algoritmi ottenendo un senso biologico interpretabile dai medici per comprendere meglio, ad esempio, lo sviluppo di gravi malattie o la loro dipendenza da fattori ambientali.
Testo redatto su fonti Politecnico di Milano del 12 maggio 2016 e del 21 giugno 2018
Per approfondimenti su GeCo: www.bioinformatics.deib.polimi.it/geco
Per approfondimenti su GMQL: www.bioinformatics.deib.polimi.it/genomic_computing
Image credit: YMGERMAN/FOTOLIA.COM
© Copyright ADEPRON – Riproduzione riservata

 BIOLOGIA COMPUTAZIONALE
BIOLOGIA COMPUTAZIONALE
INSIST e InSilc: progetti per testare l’efficacia di dispositivi medici, farmaci e interventi chirurgici per ictus e malattie coronariche attraverso simulazioni numeriche al computer
09.03.2018
Testo dell’articolo
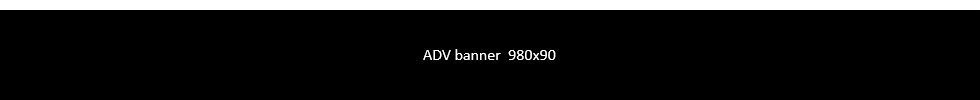
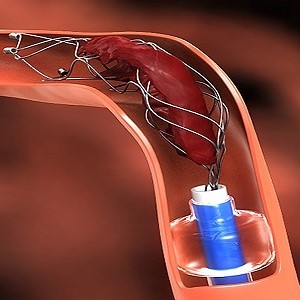
Testare l’efficacia di dispositivi medici, farmaci e interventi chirurgici per ictus e malattie coronariche attraverso simulazioni numeriche al computer, diminuendo i margini di insuccesso e riducendo drasticamente le sperimentazioni sugli animali. É l’obiettivo di due progetti di cui il Politecnico di Milano è partner, finanziati complessivamente dal Programma Horizon 2020 con 11,3 milioni di euro: INSIST (IN Silico clinical trials for treatment of acute Ischemic STroke) e InSilc (InSilico Trials for drug-eluting bioabsorbable vascular scaffold (BVS) development and evaluation). I progetti dureranno rispettivamente 3 e 4 anni e vedono la collaborazione di prestigiosi partner accademici, industriali e clinici europei.
Con l’espressione “In Silico Clinical Trials” si intende l’utilizzo della simulazione numerica al computer per sviluppare o per valutare le prestazioni di un dispositivo medico (per esempio una valvola cardiaca), di un farmaco o di una procedura di intervento chirurgico. L’ictus e le malattie coronariche sono fra le principali cause di morte nei Paesi industrializzati. Per quanto il loro trattamento sia ampiamente migliorato negli ultimi decenni, alcune situazioni restano tuttora prive di soluzioni convincenti e affidabili. L’utilizzo della realtà virtuale può senz’altro superare queste difficoltà e rendere più sicuri i trattamenti.
Il metodo seguito nei due progetti, che saranno entrambi condotti nel Laboratorio di Meccanica delle Strutture biologiche (LaBS) del Dipartimento di Chimica, Materiali e Ingegneria chimica “Giulio Natta” del Politecnico di Milano, si pone come uno tra i più innovativi e promettenti. Al LaBS saranno infatti realizzati dei modelli al computer basati sulle immagini mediche utilizzate di routine negli ospedali, in grado di rappresentare in modo dettagliato l’anatomia vascolare di pazienti colpiti da ictus o malattie del cuore. Tali modelli saranno poi utilizzati per simulare uno o più trattamenti possibili per i pazienti, al fine di prevederne l’impatto e l’evoluzione, di valutarne in anticipo il successo o il fallimento ed eventualmente per studiare le opportune modifiche.
Testo redatto su fonte Politecnico di Milano del 7 marzo 2018
Per approfondimenti sui progetti: INSIST – InSilc
Images credit: INSIST consortium
© Copyright ADEPRON – Riproduzione riservata

 NEUROSCIENZE COMPUTAZIONALI
NEUROSCIENZE COMPUTAZIONALI
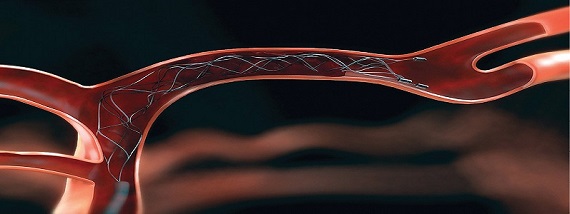
Nell’ambito del Blue Brain Project (BBP) è stato ricostruito, utilizzando un supercomputer, il primo modello digitale 3D di una piccola porzione di corteccia cerebrale di ratto
09.10.2015
Testo dell’articolo
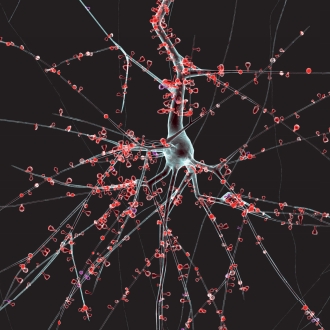
Il modello simula un volume di 0,29 mm3 di microcircuiti neurocorticali composto da 31.346 neuroni di 55 tipi morfologici diversi, con circa 40 milioni di connessioni sinaptiche. Per la simulazione è stato utilizzato il Blue Brain 4, un Power Systems HPC (High Performance Computing) installato presso il CSCS (Swiss National Supercomputing Center) a Lugano (Svizzera), una unità del Swiss Federal Institute of Technology di Zurigo (ETH Zurich). Nello specifico, si tratta del supercomputer IBM Blue Gene/Q, un sistema che, dotato di 65.536 core con architettura PowerPC A2 da 1,6 GHz, con 128 TB di memoria flash e 64 TB di memoria DRAM, è in grado di fornire una prestazione di picco di 839 TFlop/s. Si consideri che la simulazione dell’intero modello del cervello di un ratto, allo stesso livello di dettaglio, necessiterebbe di rappresentare fino a 200 milioni di neuroni, richiedendo circa 10.000 volte più memoria.
Sebbene il risultato ottenuto sia stato straordinario, è però solo il primo passo verso un obiettivo più ambizioso. Il cervello umano si comporta come un computer super performante, energeticamente autonomo, capace di ripararsi da solo e di auto apprendere. La scienza, allo stato attuale, ha compreso moltissimi aspetti del funzionamento del cervello, e dall’altra parte ha sviluppato potenti strumenti informatici di elaborazione. Il concetto che sta alla base del BBP è che, mettendo insieme le conoscenze neurologiche che i ricercatori hanno acquisito sul funzionamento di molecole cerebrali, cellule nervose e circuiti neuronali con sistemi di calcolo ad alte prestazioni (supercomputing), si può immaginare di realizzare un simulatore biologicamente molto dettagliato dell’intera struttura del cervello umano. Le ricerche nel settore sono già piuttosto avanzate, grazie agli elevati livelli tecnologici oggi disponibili e alla solida base costituita dai precedenti studi di Markram.
Con questa finalità è nato il BBP, il cui obiettivo finale è quello di riuscire a costruire, entro il 2023, una mappa completa dei microcircuiti, realizzando un modello dei 100 miliardi di neuroni del cervello umano da implementare all’interno di un supercomputer. Le modellazioni digitali offrono un approccio radicalmente nuovo per la comprensione della struttura e il funzionamento del cervello. Il metodo di ricerca del BBP sfrutta le interdipendenze tra i dati sperimentali per ottenere mappe dense del cervello, senza necessità di conoscere ogni specifico aspetto dei suoi molteplici livelli di organizzazione (molecole, cellule, micro-circuiti, regioni del cervello, l’intero cervello). Questa strategia consente di ottenere ricostruzioni digitali (modelli informatici) del cervello ad un livello di dettaglio biologico senza precedenti. Raggiungere questo risultato consentirebbe poi di trovare nuove terapie per le principali malattie degenerative delle cellule celebrali, come Alzheimer e Parkinson, ma anche di altri meccanismi, come la schizofrenia, ma questi son gli obiettivi del più generale Human Brain Project (HBP).
Testo redatto su fonte EPFL dell’8 ottobre 2015
Per approfondimenti: Reconstruction and Simulation of Neocortical Microcircuitry, DOI: 10.1016/j.cell.2015.09.029 – Cell | 08.10.2015
Blue Brain Project: bluebrain.epfl.ch
Human Brain Project: www.humanbrainproject.eu
Image credit: EPFL/BBP
© Copyright ADEPRON – Riproduzione riservata

 BIOLOGIA COMPUTAZIONALE
BIOLOGIA COMPUTAZIONALE
Sviluppato un modello computazionale che ricostruisce il meccanismo con il quale il sistema nervoso riesce a rigenerarsi e a ripristinare le connessioni fra i neuroni
25.06.2015
Testo dell’articolo
L’utilizzo di modelli computazionali può aiutare a comprendere molti aspetti della crescita degli assoni: essi si estendono durante lo sviluppo del sistema nervoso e, alla loro estremità, si trova il cono di accrescimento, il sistema di navigazione biologico che percepisce gli stimoli extracellulari e che ne direziona l’avanzamento. Come un autentico navigatore, il cono di accrescimento recepisce i segnali necessari al direzionamento attraverso strutture sensoriali specializzate (chiamate “filopodia”) e “traduce” questi segnali in movimenti specifici, mediante complesse reazioni chimiche. Il modello sviluppato, presentato su “Scientific Reports” in “A hybrid computational model to predict chemotactic guidance of growth cones“, è in grado di riprodurre in maniera fedele tutti questi aspetti della crescita assonale.
Per gli autori della ricerca, l’obiettivo di molti neuroscienziati e bioingegneri che lavorano nel campo della rigenerazione dei tessuti nervosi periferici e centrali (per esempio dei nervi periferici e del midollo spinale) è comprendere i meccanismi con cui gli assoni si orientano durante la crescita è da sempre. Queste conoscenze potrebbero consentire lo sviluppo di nuovi e più efficaci sistemi artificiali (come le “neuroprotesi” per la rigenerazione neuronale), basati sul controllo della crescita degli assoni, apportando notevoli benefici nel trattamento dei danni, derivanti da traumi o patologie, a carico del sistema nervoso.
Testo redatto su fonte Scuola Superiore Sant’Anna di Pisa del 18 giugno 2015
Per approfondimenti: A hybrid computational model to predict chemotactic guidance of growth cones, DOI: 10.1038/srep11340 – Scientific Reports | 18.06.2015
Image credit: Scientific Reports/Nature (2015) DOI: 10.1038/srep11340
© Copyright ADEPRON – Riproduzione riservata

 FLUIDODINAMICA CARDIACA
FLUIDODINAMICA CARDIACA
Un innovativo approccio interdisciplinare per identificare precocemente il rischio di scompensi cardiaci apre la strada ai futuri processi diagnostici e di ottimizzazione chirurgica
22.10.2014
Testo dell’articolo
L’integrazione con i metodi ingegneristici può aiutare a comprendere meglio questo fenomeno clinico in quanto il flusso del sangue all’interno del ventricolo sinistro è caratterizzato dalla presenza di un vortice che guida il moto lungo traiettorie curve percorrendo l’intera cavità ventricolare. Dal punto di vista dinamico, la peculiarità di questo processo consiste nella presenza di elevate accelerazioni, e quindi forze emodinamiche, dovute a rapide variazioni nella direzione e nell’intensità della velocità del sangue. Gli attuali modelli clinici non tengono in alcun conto della presenza del sangue all’interno della camera cardiaca. Il sangue è un materiale incomprimibile, con lo stesso peso del tessuto, pertanto esso mette in contatto i diversi segmenti del miocardio attraverso la colonna di fluido tra di essi. In presenza di un flusso vorticoso intraventricolare, la comunicazione tra regioni lontane viene modulata attraverso il movimento del sangue, cosicché la rapida accelerazione-decelerazione in prossimità di una regione può dar luogo a sollecitazioni in un’altra.
È stato dimostrato di recente che lo sviluppo fisiologico del cuore allo stato embrionale è guidato dalle azioni emodinamiche (shear stress) che stimolano la moltiplicazione cellulare. Nella stessa prospettiva è naturale ipotizzare che le azioni emodinamiche possano stimolare anche nel cuore adulto le variazioni geometriche associate con lo scompenso cardiaco. In numerose patologie, la sequenza di contrazione e rilasciamento dei segmenti miocardici perde la naturale sincronia e porta allo sviluppo di un errato flusso vorticoso intraventricolare. Quest’ultimo genera un disallineamento dello scambio dinamico di forze tra sangue e tessuto e porta alla nascita di sollecitazioni su regioni errate e in momenti inopportuni durante il ciclo cardiaco. Per esempio, se un segmento si contrae anticipatamente rispetto a un altro, la forza generata dal primo imprime una spinta sul secondo, ancora inattivo, anziché essere sfruttata per l’eiezione in aorta; parimenti un’iniziale rigidità di un segmento durante il rilasciamento provoca la deviazione del flusso in ingresso verso un’altra regione che viene così sottoposta a un inatteso incremento di carico. La nascita di sollecitazioni anomale suscita un adattamento fisiologico, per esempio attraverso meccanismi di trasduzione meccanica che inducono processi di genomica e proteomica, con il conseguente sviluppo d’ipertrofia e apoptosi regionale che avvia il processo di rimodellamento.
L’integrazione ingegneristica alla medicina conduce verso la comprensione del ruolo epigenetico della fluidodinamica cardiaca al fine di permettere un’identificazione precoce del rischio di scompenso destinata a divenire parte integrante dei futuri processi diagnostici nonché di ottimizzazione chirurgica.
Testo redatto su fonte Università di Trieste del 21 ottobre 2014
Per approfondimenti: The vortex—an early predictor of cardiovascular outcome? – Nature Reviews Cardiology | 03.06.2014
Image credit: Gianni Pedrizzetti, Università di Trieste
© Copyright ADEPRON – Riproduzione riservata

 GENOMICA COMPUTAZIONALE
GENOMICA COMPUTAZIONALE
SISSA: pubblicata una rassegna sistematica delle metodologie delle ricerche sperimentali sul DNA attraverso 25 anni di simulazioni al computer
03.01.2014
Testo dell’articolo
********************************************
Il DNA svolge le sue attività “disciolto” nel nucleo delle cellule. In questo stato sintetizza le proteine e, anche se assomiglia a un gomitolo arruffato, in realtà la sua struttura rispetta regole precise che sono importanti per lo svolgimento delle sue funzioni. I biologi hanno studiato il DNA osservandolo sperimentalmente, con varie tecniche, e solo in anni recenti a queste metodologie sperimentali si è affiancata la ricerca “in silico”, vale a dire lo studio del DNA attraverso le simulazioni al computer.
È un campo di studi recente, ma ha già dato un importante contributo alle conoscenze in questo settore. Angelo Rosa, fisico teorico della SISSA, con la collaborazione di Christophe Zimmer, fisico sperimentale dell’Istituto Pasteur di Parigi, ha fatto il punto sullo stato dell’arte in questo nuovo ma ricco approccio, con una rassegna sistematica appena pubblicata sulla rivista “International Review of Cell and Molecular Biology”. “A parte qualche rara eccezione, abbiamo ricompilato praticamente tutti i modelli apparsi finora”, spiega Rosa. “Si tratta di una rassegna destinata ai biologi, in quanto limita al massimo l’uso di formule matematiche che appesantiscono la lettura. Credo si tratti del primo lavoro di questo tipo. Il lavoro è in realtà interessante anche per i fisici e matematici che, si avvicinano a questo campo così nuovo”.
I due fisici hanno ripercorso 25 anni di modelli al computer: “in questo periodo relativamente breve i modelli sono diventati molto più raffinati, e questo grazie allo sviluppo dei computer” spiega Rosa. “Oggi sono possibili simulazioni molto più dettagliate e predittive, che permettono quindi di orientare il lavoro degli sperimentali in direzioni fino a pochi anni fa impensate”. “Si tratta di uno strumento utile che senza addentrarsi nel dettaglio matematico offre al biologo una panoramica su studi che via via si affiancheranno sempre più spesso a quelli più tradizionali” continua Rosa. ”Già oggi per esempio esistono software che partendo dai dati sperimentali permettono di ricostruire la struttura di parti specifiche dei cromosomi. Penso che se i computer continueranno a evolvere come hanno fatto finora – e non c’è motivo di pensare il contrario – saremo in grado di ricostruire interi cromosomi”.
“Al momento due sono le prospettive future della ricerca in silico sul DNA nucleare”, conclude Rosa, “capire nel dettaglio la meccanica dell’espressione genica (i dettagli della sintesi delle proteine) e identificare con precisione dove sono i cromosomi quando il DNA è sbrogliato nel nucleo”.
Testo redatto su fonte SISSA del 3 gennaio 2014
Image credit: SISSA
© Copyright ADEPRON – Riproduzione riservata

 NEUROSCIENZE COMPUTAZIONALI
NEUROSCIENZE COMPUTAZIONALI
“Human Brain Project”: il progetto di ricerca finanziato dalla UE con 1,2 miliardi di euro per simulare con un supercomputer l’intero cervello umano entro il 2020
26.09.2013
Testo dell’articolo
Il progetto si è aggiudicato da parte della Commissione Europea un finanziamento complessivo di circa 1,2 miliardi di euro in dieci anni, dei quali 54 milioni di euro per 30 mesi copriranno la fase di lancio. Al progetto – coordinato dal neuroscienziato Henry Markram dell’Ecole Polytechnique Fédérale di Losanna – partecipano 87 istituti di ricerca di 24 paesi del mondo. Di essi cinque sono italiani: il Laboratorio Europeo di Spettroscopia Non lineare dell’Università di Firenze (LENS/UNIFI), il Politecnico di Torino (POLITO), l’Università di Pavia (UNIPV), l’Istituto di Ricovero e Cura a Carattere Scientifico Fatebenefratelli (IRCCS Fatebenefratelli) di Brescia e il Consorzio Interuniversitario CINECA, il maggior centro di supercalcolo in Italia.
Le tecnologie informatiche (ICT) sono al centro del progetto: i dati clinici, raccolti dal tutto il mondo, su innovative piattaforme bioinformatiche, permetteranno ai ricercatori di decifrare le informazioni più utili dal punto di vista clinico e inserirle in un modello virtuale delle malattie. L’obiettivo è costruire un simulatore dell’intera attività del cervello umano, un modello con cento miliardi di neuroni che consentirebbe di comprendere malattie neurodegenerative e altri disturbi del sistema nervoso (Alzheimer, Parkinson, epilessia e schizofrenia), per sviluppare tecniche per una diagnosi oggettiva delle affezioni del cervello, comprendere i meccanismi ad esse sottesi e mettere a punto efficaci terapie e nuovi farmaci.
Il compito dei partner italiani sarà quello di realizzare le immagini tridimensionali del cervello (LENS/UNIFI), immagazzinarle in un grosso computer per processarle a distanza (CINECA), simulare il funzionamento delle connessioni celebrali (UNIPV), trattare una grossa quantità di dati clinici e medici (IRCCS Fatebenefratelli) e realizzare strutture elettroniche che simulino i neuroni (POLITO).
La ricerca tuttavia non punta solo a simulare il funzionamento del cervello, ma si propone anche di creare una visione unitaria di questo organo così complesso. Connettendo fra di loro le conoscenze neurologiche sul funzionamento delle molecole celebrali, delle cellule e dei i circuiti neuronali con quelle sui più potenti database attualmente sviluppati grazie alle tecnologie ICT, si può arrivare a costruire un simulatore biologicamente molto dettagliato dell’intera attività del cervello umano, ma anche a porre le basi per la creazione delle nuove generazioni di supercomputer intelligenti, e mettere tutto questo a disposizione della comunità scientifica internazionale.
Per approfondimenti sull’HBP: www.humanbrainproject.eu
Image credit: Laboratory of Neuro Imaging and Martinos Center for Biomedical Imaging, Consortium of the Human Connectome Project
© Copyright ADEPRON – Riproduzione riservata